2. 东北林业大学盐碱地生物资源环境研究中心, 哈尔滨, 150040
3. 山西省农科院经济作物研究所, 汾阳, 032200
4. 海南省农作物分子育种重点实验室, 海南省热带农业资源开发利用研究所, 三亚, 572025
 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2012 年, 第 10 卷, 第 55 篇 doi: 10.5376/mpb.cn.2012.10.0055
收稿日期: 2012年12月06日 接受日期: 2012年12月07日 发表日期: 2012年12月14日
Luo et al., 2012, QTL Mapping and Analysis of Traits Related to Pod Dehiscence in Soybean, Legume Genomics and Genetics, Vol.3, No.3 14-20 (doi: 10.5376/lgg. 2012.03.0003)
大豆豆荚开裂(pod dehiscence)是大豆重要的生育特性,而在生产上豆荚开裂则是影响大豆产量的重要不利因素之一。本研究利用大豆重组自交系群体(JINF群体)的112个株系,采用复合区间作图法(composite interval mapping, CIM)法,对大豆豆荚开裂性状(PDH),豆荚厚宽比(RTW)以及完熟天数(DFM) 3个性状进行相关性分析及QTL定位。研究结果表明,PDH与RTW之间呈现出极显著的负相关性(P=0.01, -0.960),与DFM呈显著负相关性(P=0.05, -0.740);而RTW和DFM之间呈现显著正相关性(P=0.05, 0.867)。QTL定位分析表明,在C2连锁群(即第6染色体)上检测到一个控制豆荚开裂的QTL座位,命名为qPDH6-1连锁,位于标记Satt062和Satt520之间,LOD值为12.15,遗传距离为3.1 cM,可解释的表型变异率为49.44%。对于完熟天数,我们检测3个与之连锁的QTL座位,分别命名为qDFM6-1、qDFM6-2和qDFM18-1,分别位于标记Sat_062和Satt520、Satt291和Satt305及Satt217和Satt130之间,LOD值分别为6.59、2.85和2.81,遗传距离为7.2 cM、12.3 cM和0.6 cM,可解释的表型变异率为21.77%、9.74%及7.74%。其中qDFM6-1座位与qPDH6-1位于相同的标记区间(Satt062和Satt520之间);qDFM6-2和qDFM18-1分别位于第6和第18染色体的Satt291和Satt305之间以及Satt217和Satt130之间。我们还检测到3个与大豆豆荚厚宽比连锁的QTL座位,分别位于第11、4和8染色体上,命名为qRTW11-1、qRTW4-1和qRTW8-1,分别位于标记Satt426和Satt509、Satt361和Satt399及Satt187和Satt377之间,LOD值分别为3.00、2.77和2.14,遗传距离分别为10.0 cM、0.4 cM及3.7 cM,可解释的表型变异率为9.03%、8.20%和7.02%。
大豆(Glycine max L.)豆荚开裂(pod dehiscence, PDH)是指大豆在成熟期,豆荚干燥失水之后,豆荚沿着背部、腹部的缝合线裂开,然后散播出种子的特性。这种开裂特性是为了更好的繁衍后代经过不断进化形成的,在野生大豆中这种特性仍然是非常普遍的。野生大豆在向栽培大豆的驯化过程中,人类在不断的淘汰这种豆荚开裂特性,以便有效的收获大豆籽粒。尽管豆荚不开裂或者不易开裂是培育大豆栽培品种的育种目标之一,但是仍然有许多大豆品种在延迟收割或天气干燥的情况下,豆荚会自然裂开。这在大豆生产中会导致大豆产量的损失。
已有科学家采用大豆豆荚的开裂抗性和感性的亲本品种,构建出重组自交系(recombinant inbred lines, RILs),并且利用这些家系对豆荚开裂特性进行QTL定位和分析。Bailey等(1997)在大豆F4代家系中定位到12个与豆荚开裂特性相关RFLP (Restriion fragment length polymorphism)标记,这些QTL座位分别位于第2、第15、第16和第19染色体上。Funatsuki等(2006)利用复合区间作图法,在大豆重组自交系中定位到一个大豆豆荚开裂特性相关的QTL,将其命名为qPDH1,该QTL座位位于第16染色体上的SSR标记Sat_093和Sat_366之间,并估计出该QTL的遗传距离为2.9 cM;2008年,他们利用4个不同遗传背景的大豆材料,再次证明了控制大豆豆荚开裂的QTL座位qPDH1位于Sat_093和Sat_366之间,表型变异率为50%,而且还发现其中控制豆荚开裂的等位基因,在不同的遗传背景下具有多重位点(Funatsuki et al., 2008)。Suzuki等(2009)认为qPDH1座位主要控制大豆豆荚的开裂过程,而大豆的形态学特征在该过程中并没有发挥明显的作用。Yamada等(2009)通过对几个大豆群体进行分析,发现控制豆荚开裂抗性的基因在不同的遗传背景下具有一定的差异,但是豆荚开裂特性仍然是由qPDH1座位控制的。Kang等(2009)定位到一个主要的以及3个次要的QTL座位,该主要QTL座位位于第16染色体上,也位于qPDH1附近;3个次要的QTL座位分别位于第5、第14和第10染色体上,并且认为大豆的豆荚开裂特性在不同的遗传背景下具有一定的差异性。
在本研究中,我们利用本实验室和山西省农科院共同构建的大豆重组自交系群体(JINF群体) (刘学义等, 2003),对大豆豆荚的开裂特性、以及与之可能关联的两个性状即大豆的完熟天数(dys of full maturety, DFM)和豆荚厚/宽比(rtio of thichness to width, RTW),进行相关分析和QTL定位以期深入了解大豆豆荚开裂特性以及与相关性状的相互关系,为进一步克隆基因以及分子育种应用奠定基础。
1结果分析
1.1三个性状之间的相关性分析
对大豆JINF群体中的112个株系的大豆豆荚的开裂特性(pod dehiscence, PDH)、大豆的完熟天数(days of full maturity, DFM)及豆荚厚宽比(ratio of thickness to width, RTW)进行表型测定和统计后,用SPSS 16.0软件并对上述3个性状进行相关性分析。结果显示大豆豆荚的开裂特性与其它两个性状(豆荚的厚/宽比, 大豆的完熟天数)之间具有一定的负相关性。其中,PDH与RTW便有极显著的负相关性(P=0.01, -0.960);PDH与DFM呈现显著负相关(P=0.05, -0.740)。而RTW与DFM之间则呈现显著正相关(P=0.05, 0.867)。
1.2大豆豆荚开裂性状的QTL定位
对大豆JINF群体中112个株系的豆荚开裂性状进行QTL定位分析,利用WinQTLCart 2.5软件中的复合区间作图法(composite interval mapping, CIM)设定LOD值>2.0,定位到该性状在连锁群C2 (第6染色体)上有一个QTL座位,位于标记Sat_062和Satt520之间,命名为qPDH6-1,LOD值达到12.15,遗传距离为3.1 cM,可解释的表型变异率为49.44% (图1, 表1)。
.jpg) 表1 对大豆JINF群体的112个株系中, 3个与豆荚开裂特性相关性状定位到的SSR标记
Table 2 Putative independent SSR markers associated with QTLs for the traits related to the PD in the 112 plants of JINF population |
 图1 大豆豆荚开裂性状QTL在第6染色体上的位置
Figure 1 Location of the QTL for PDH on chromosome 6 in the JINF RIL population |
1.3大豆豆荚的厚宽比的QTL定位
我们利用WinQTLCart 2.5软件的复合区间作图法(CIM法)对大豆JINF群体的112个株系的豆荚厚宽比进行定位分析,设定LOD值>2.0,共检测到3个QTL座位,分别命名为qRTW11-1、qRTW4-1和qRTW8-1 (图2, 表1)。
图2 大豆厚宽比性状的3个QTL在第11, 第4和第8染色体上的位置 Figure 2 Location of three QTLs for the ratio of thickness to width on the chromosome 11, 4 and 8 in the JINF RIL population |
qRTW11-1位于连锁群B1 (第11染色体)上的标记Satt426和Satt509之间,LOD值为3.00,可解释的表型变异率为9.03%,遗传距离为10.0 cM;qRTW4-1位于连锁群C1 (第4染色体)上的标记Satt361和Satt399之间,LOD值为2.77,可解释的表型变异率为8.20%,遗传距离0.4 cM;qRTW8-1位于连锁群A2 (第8染色体)上的标记Satt187和Satt377之间,LOD值为2.14,可解释的表型变异率为7.02%,遗传距离3.7 cM。
1.4大豆完熟天数的QTL定位和分析
采用WinQTLCart 2.5软件的复合区间作图法(CIM法),对大豆JINF群体中的112个株系进行完熟天数的QTL定位分析,设定LOD值>2.0,共检测到3个QTL座位,分别命名为qDFM6-1、qDFM6-2以及qDFM18-1 (图3, 表1)。
.png) 图3 大豆完熟天数的QTL在第6和第18染色体上的位置 Figure 3 Location of the QTLs for the trait of the days of full maturity on the chromosome 6 and 18 in the JINF RIL population |
qDFM6-1和qDFM6-2座位都位于连锁群C2 (第6染色体)上,分别位于标记Sat_062和Satt520之间以及Satt291和Satt305之间,LOD值分别为6.59和2.85,可解释的表型变异率分别为21.77%、9.74%,遗传距离分别为7.2 cM和12.3 cM;qDFM18-1座位则位于连锁群G (第18染色体)上Satt217和Satt130标记之间,LOD值为2.81,可解释的表型变异率为7.74%,遗传距离为0.6 cM。
1.5相关QTL座位的基因组信息分析
根据大豆基因组网站(http://www.phytozome.net/cgi-bin/gbrowse/soybean/)上的基因组注释信息,我们发现qPDH6-1座位附近有一个具有生长素反应蛋白活性的基因Glyma06g00860.1,以及一个编码水解酶活性的基因Glyma06g01500.1。在qRTW4-1座位附近发现了一个编码生长素转移蛋白基因活性的Glyma04g43150.1,以及一个编码多聚半乳糖醛酸酶的基因Glyma04g43340.1。在qDFM18-1座位附近发现了一个编码多聚半乳糖醛酸酶的基因Glyma18g16870.1。
2讨论
在以往的研究中,都认为影响大豆豆荚开裂特性的主效QTL (qPDH1座位)位于第16染色体上(Funatsuki et al., 2006; 2008; Liu et al., 2007; Kang et al., 2009; Suzuki et al., 2010)。Kang等(2009)的研究认为在不同的大豆品种中,豆荚的开裂程度具有一定的差异。我们的研究将大豆豆荚开裂性状的QTL (qPDH6-1)定位在第6染色体上,这与已有的报道都不一样。这可能与我们采用高代重组自交系群体以及驯化程度低的农家种“灰布支”有关。
在我们的定位结果中,发现在C2连锁群(第6染色体)上具有多个QTL座位。其中,PDH的一个QTL座位qPDH6-1在第6染色体上的0.4~3.6 cM的位置、DFM的主效QTL座位qTFM6-1的位置为0.1~7.3 cM,并且这两个QTL都位于Satt_062和Sa- tt520标记之间。而与DFM连锁的qTFM6-2座位在第6染色体上的位置为16.4~28.7 cM,这3个QTL座位重叠或相邻,这表明在该染色体上,具有控制大豆豆荚开裂相关性状的重要基因。
Tsuchiya (1987)的研究表明大豆豆荚的厚度和宽度之间的比值对其开裂特性有一定的影响,同时认为,大豆豆荚的厚度和宽度在大豆的豆荚开裂特性的抗性和感性品种之间,并没有明显的差异。Caviness (1969)认为大豆豆荚的开裂特性还受大豆籽粒大小的影响,而大豆籽粒的大小也影响着大豆豆荚的厚宽比的大小。本研究认为大豆豆荚的开裂特性和豆荚的厚宽比及完熟时间均呈现显著的负相关性,证实了前人的研究结论。
Arnaud等(2010)认为生长素在双子叶植物的豆荚的开裂过程中具有重要的作用。而我们通过对该特性所定位到的连锁QTL座位进行生物信息学分析时,在第6染色体上的qPDH6-1座位的附近发现了1个具有生长素反应蛋白活性的基因Glyma06g00860.1,而在第4染色体上于豆荚厚宽比连锁的qRTW4-1座位附近也有一个生长素转移蛋白基因Glyma04g43150.1,尽管目前对于这2个基因的详细注释并不清楚,但是我们的发现也从一定的程度上认为生长素在大豆豆荚开裂过程中可能具有重要的作用,为下一步的研究提供了一定的方向。
Christiansen等(2002)在大豆中发现多聚半乳糖醛酸酶(polygalacturonase, PG)与油菜中的该基因具有一定的差异,但是Petersen等(1996)等还是认为油菜荚果的开裂特性与多聚半乳糖醛酸酶相关,巧合的是,我们在qRTW4-1座位附近发现了Glyma04g43340.1以及第18染色体上的qDFM18-1座位附近的Glyma18g16870.1基因编码的蛋白都具有多聚半乳糖醛酸酶活性,这说明PG可能在大豆的开裂过程中也具有一定的作用,也为下一步的研究提供一定的方向。
水解酶在荚果植物的开裂过程中起着关键的作用,这在荚果的开裂特性的研究中是被普遍接受的(Fry et al., 1992; Agrawal et al., 2002)。而在第6染色体qPDH6-1座位附近,我们发现了一个编码水解酶的基因Glyma06g01500.1。
尽管对于上述基因的详细注释还不够清楚,以及这些基因具有的相应活性是否与荚果的开裂相关的性状有关,但是我们的研究表明生长素、相应的多聚半乳糖醛酸酶以及水解酶在大豆豆荚开裂特性的调控网络中可能具有的一定的作用。此外,Arnaud等(2011)等还认为在拟南芥中,由启动子区域的基因序列来对荚果的开裂特性进行调控,这为下一步荚果开裂特性的调控网络和基因的具体研究提供了重要的研究材料和方向。
3材料与方法
3.1供试材料
大豆母本:栽培品种“晋豆23”(Glycine max Merr., CV. Jinbean 23),在大豆成熟之后豆荚不容易裂开;父本:半野生品种“灰布支”(Glycine max Merr., CV. ZDD2315),大豆成熟之后,荚果因为失水容而易开裂;以及由它们构建而成的纯合的重组自交系(recombinant inbred lines, RILs)体系(JINF群体)112个株系(刘学义等, 2003)均由山西省农科院经济作物研究所刘学义研究员提供。
3.2大豆遗传图谱及QTL分析
宛煜嵩等(2005)利用大豆JINF群体为材料,整合了一张具有227个SSRs标记的大豆遗传连锁图,后来巩鹏涛等(2006)基于该研究中的SSRs标记以及大豆公共图谱中的标记,重新整合了一张新的图谱。本研究所使用的遗传图谱是基于巩鹏涛等(2006)整合的图谱中的240个SSRs标记组成的大豆分子遗传图谱。
在SPSS 16.0软件Analyze下的Descriptive Sta- tistics中选择Frequencies,打开频数统计对话框后,在Charts中选择Histogram选项及With normal curve后进行各性状的柱形图的正态分布统计。在SPSS 16.0软件Analyze下的Correlate中选择Bivariate,然后在Options中选中Means and standard dev- iations以及Excludes cases pairwise,再设置Correlation Coeffecients选项为Pearson,Test of Signifi- cance中选择Two-tailed,然后选中Flag significant correlations之后进行3个性状之间的相关性分析。各个性状的QTL定位是在WinQTLCat 2.5软件(http://statgen.ncsu.edu/qtlcart/WQTLCart.htm)中导入数据后,采用CIM法,将Walk speed设定为2,然后将By Manual Input设定为11.50,其余为默认值。
3.3田间种植
供试材料在播种前进行晒种1 d,于2011年8月5日在海南省热带农业资源开发与利用研究所的防虫网室中进行播种。播种采用20 cm×20 cm的方盆进行,每盆装泥土4 kg (泥土也同种子一起进行翻晒一天),播种前在泥土中拌上有机肥。播种时每个亲本(晋豆23×灰布支)各3盆,112个RILs各1盆,每盆播种5~6粒种子,待苗长至V4期(第1个三出复叶节至第2个三出复叶节)进行定苗,每盆保留3个株系。在苗生长期进行定期的浇水及除草等相关的管理工作,并在相应的生长期做好数据记录和采收工作。
3.4表型测定
3.4.1豆荚开裂性状的表型评估
待大豆长至R8期(Full maturity, 即有95%的豆荚的颜色变为黄色、棕色或者黑色)时,便采集荚果,每个植株采摘10~20个不等,并且在室温环境下干燥15 d。
然后进行热处理,具体的处理方法参照Tukamuhabwa等(2002)的研究进行,并有所改动,即将烘箱预热至80℃后,放入荚果烘5 h (为了防止豆荚爆裂影响结果的统计,将荚果放入三角瓶后再放入烘箱),然后取出三角瓶统计开裂豆荚的个数,并记录其占总荚果的百分比,该百分比即为该大豆植株的开裂程度,然后在每个株系的3个重复中求平均值,利用SPSS 16.0获得其柱形分布图(图4)。
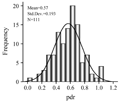 图4 112个大豆株系中豆荚开裂性状表型评估的分布及标准曲线 Figure 4 The frequency distribution in 112 soybean plants for the shattering rate |
3.4.2豆荚厚宽比的表型评估
在大豆R7期(Beginning maturity)时,采集每个株系的荚果10个,分别测量荚果的总宽度和总厚度。
大豆荚宽的测量参照刘学义等(2003)的测量方法,即将10个荚果按宽度方向连续排列之后,量取总长度,然后求平均值。
大豆荚厚的测量是仿照上述方法,由笔者制造一个简易装置,使得荚果能够按照其厚度的方向,向上依次叠加,然后再量取总厚度,之后求平均值。
然后求出每个株系豆荚的厚/宽比,再在每3个重复中求平均值,并用SPPS 16.0统计其分布图(图5)。
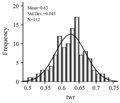 图5 112个大豆株系中豆荚厚宽比的分布及标准曲线 Figure 5 The frequency distribution in 112 soybean plants for the rate of width and thickness of pods (T/W) |
3.4.3大豆完熟天数的表型评估
每个株系从种植到R8期的天数即为大豆成熟所需要的天数,并在每3个重复中求平均值,SPSS 16.0统计其柱形分布图(图6)。
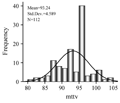 图6 112个大豆株系完熟天数的分布及标准曲线 Figure 6 The frequency distribution in 112 soybean plants for the days of full maturity |
作者贡献
罗汝叶完成本研究的试验设计和实施,文章初稿的撰写和内容的修改。巩鹏涛负责本研究的设计,科研及写作方面的指导,数据分析以及论文写作的修改。刘学义参与本研究的方案制定和材料应用方面的指导。赵德刚是项目主持人。
致谢
本研究由国家科技支撑项目资助(2007BAD59B05)。作者感谢海南省热带农业资源开发利用研究所方宣钧博士对本文的指导和对论文的审读;感谢省资源所技术员黎仲刚对试验材料的种植和田间管理;两位匿名的同行评审人的评审建议和修改建议、阅读和审稿。
参考文献
Agrawal A.P., Basarkar P.W., Salimath P.M., and Patil S.A., 2002, Role of cell wall-degrading enzymes in podshatt-ering process of soybean, Glycine max (L.) Merrill, Current Science, 82(1): 58-61
Arnaud N., Girin T., Sorefan K., Fuentes S., Wood T.A., La- wrenson T., Sablowski R., and Ostergaard L., 2010, Gi- bberellins control fruit patterning in Arabidopsis thaliana, Genes and Development, 24 (19): 2127-2132
http://dx.doi.org/10.1101/gad.593410 PMid:20889713 PMCid:2947765|
Arnaud N., Lawrenson T., Østergaard L., and Sablowski R., 2011, The same regulatory point mutation changed seed- dispersal structures in evolution and domestication, Curr. Biol., 21: 1215-1219
http://dx.doi.org/10.1016/j.cub.2011.06.008 PMid:21737279
Bailey M.A., Mian M.A.R., Carter T.E., Ashley D.A. and Boer- ma H.R., 1997., Pod dehiscence of soybean: Identification of quantitative trait loci, The Journal of Heredity 88: 152-154
http://dx.doi.org/10.1093/oxfordjournals.jhered.a023075
Caviness C.E., 1969, Heritability of pod dehiscence and its association with some agronomic characters in soybeans, Crop Science, 9(2): 207-209
http://dx.doi.org/10.2135/cropsci1969.0011183X000900020029x
Christiansen L.C., Dal Degan F., Ulvskov P., and Borkhardt B., 2002, Examination of the dehiscence zone in soybean pods and isolation of a dehiscence-related endopoly- galacturonase gene, Plant Cell and Environment, 25(4): 479-490
http://dx.doi.org/10.1046/j.1365-3040.2002.00839.x
Fry S., Smith R., Renwick K., Martin D., Hodge S., and Matt- hews K., 1992, Xyloglucan endotransglycosylase, a new wall-loosening enzyme activity from plants, Biochemical Journal, 282: 821-828
PMid:1554366 PMCid:1130861
Funatsuki H., Hajika M., Hagihara S., Yamada T., Tanaka Y., Tsuji H., Ishimoto M., and Fujino K., 2008, Confirmation of the location and the effects of a major QTL controlling pod dehiscence, qPDH1, in soybean, Breeding Science, 58(1): 63-69
http://dx.doi.org/10.1270/jsbbs.58.63
Funatsuki H., Ishimoto M., Tsuji H., Kawaguchi K., Hajika M., and Fujino K., 2006, Simple sequence repeat markers linked to a major QTL controlling pod shattering in soybean, Plant Breeding, 125(2): 195-197
http://dx.doi.org/10.1111/j.1439-0523.2006.01199.x
Kang S.T., Kwak M.H., Kim H.K., Choung M.G., Han W.Y., Baek I.Y., Kim M.Y., Van Kyujung, and Lee S.H., 2009, Population-specific QTLs and their different epistatic interactions for pod dehiscence in soybean [Glycine max (L.) Merr.], Euphytica 166: 15-24
http://dx.doi.org/10.1007/s10681-008-9810-6
Liu B.H., Fujita T., Yan Z.H., Sakamoto S., Xu D.H., and Jun A.B.E., 2007, QTL mapping of domestication-related traits in soyben (Glycine max), Annals of Botany, 1-12
Moncada P., Martínez C.P., Borrero J., Chatel M., Gauch Jr. H., Guimaraes E., Tohme J., and McCouch S.R., 2001, Quan- titative trait loci for yield and yield components in an Oryza saliva×Rryza rufipegon BC2F2 population evaluated in an upland environment, Theor. Appl. Genet., 102: 41-52
http://dx.doi.org/10.1007/s001220051616
Paterson A.H., Y.R., Li Z.K., Schertz K.F., Doebley J.F., Pinson S.R.M., Liu S.C., Stansel J.W., and Irvine J.E., 1995, Con- vergent domestication of cereal crops by independent mutations at corresponding genetic loci, Science, 269 (5231): 1714-1718
http://dx.doi.org/10.1126/science.269.5231.1714 PMid:17821643
Pedersen M., Sander L., Child R., van Onckelen H., Ulvskov P., and Borkhardt B., 1996, Isolation and characterization of a pod dehiscence zone-specific polygalacturonase from Brassica napus, Plant Molecular Biology, 31: 517-527
http://dx.doi.org/10.1007/BF00042225
Suzuki M., Fujino K., Nakamoto Y., Ishimoto M., and Funat- suki H., 2010, Fine mapping and development of DNA markers for the qPDH1 locus associated with pod dehiscence in soybean, Mol. Breeding, 25: 407-418
http://dx.doi.org/10.1007/s11032-009-9340-5
Tsuchiya T., 1987, Physiological and genetic analysis of pod shattering in soybean, Japan Agricultural Research Quar terly, 21: 166-175
Tukamuhabwa P., Rubaihayo P., and Dashiell K.E., 2002, Genetic components of pod shattering in soybean, Euphytica, 125 (1): 29-34
http://dx.doi.org/10.1023/A:1015711421223
Umezawa T., Sakurai T., Totoki Y., Toyoda A., Seki M., Ishiwata A., Akiyama K., Kurotani A., Yoshida T., Mochida K., Kasuga M., Todaka D., Maruyama K., Nakashima K., Enju A., Mizukado S., Ahmed S., Yoshiwara K., Harada K., Tsubokura Y., Hayashi M., Sato S., Anai T., Ishimoto M., Funatsuki H., Teraishi M., Osaki M., Shinano T., Akashi R., Sakaki Y., Yamaguchi- Shinozaki K., and Shinozaki K., 2008, Sequencing and analysis of approximately 40 000 soybean cDNA clones from a fulllength-enriched cDNA library, DNA Res., 15: 333-346
http://dx.doi.org/10.1093/dnares/dsn024 PMid: 18927222 PMCid:2608845
Yamada T., Funatsuki H., Hagihara S., Fujita S., Tanaka Y., Tsuji H., Ishimoto M., Fujino K., and Hajika M., 2009, A majior QTL, qPDH1, is commonly involved in shattering resistance of soybean cultivars, Breeding Science, 59: 435-440
http://dx.doi.org/10.1270/jsbbs.59.435
Gong Pengtao, Mu Jingui, Zhao Jinrong, Wang Xiaoling, Bai Yangnian, and Fang Xuanjun, 2006, An integrated soybean genetic linkage map comprising 315 SSRs and 40 AFLPs, Molecular Plant Breeding, 4(3): 309-316
Liu Xueyi, Wan Yusong, Wang Zhen, Ma Junkui, Shi Hong, Ren Xiaojun, Ren Donglian, Zhang Xiaohu, Li Youying, Feng Fengming, Xu Jun, Li Yongqi, and Fang Xuanjun, 2003, Development of soybean recombinant inbred lines Jinf (Jinbean23×ZDD2315) and evaluation by SSR molecular marker and agronomic traits, Molecular Plant Breeding, 1(2): 157-177
Wan Yusong, Wang Zhen, Xiao Yinghua, Lv Bei, and Fang Xuanjun, A soybean genetic linkage map comprising of 227 SSR loci in a soybean RIL population, Molecular Plant Breeding, 3(1): 15-20)